
Viren sind Krankheitserreger mit extrem engem Wirtsbereich. Ihre phylogenetische Herkunft ist ungewiß, doch schon immer wurde darüber spekuliert, daß es sich um "vagabundierende Gene" handele, die sich aus dem Wirtsorganismusgenom oder dem Genom einer verwandten Art ausgegliedert hätten. In den letzten Jahren wurde auch die Alternative in Erwägung gezogen, daß es Nebenprodukte des RNS-Processing sind. Alle Annahmen bleiben jedoch im Bereich der Hypothesen, solange keine beweiskräftigen Belege für die eine oder die andere Alternative vorgelegt werden können. Detailierte Beschreibungen aller Pflanzenviren enthält die Datenbank VIDE (Plant Viruses Online), erreichbar über das Botanik online INTERFACE:
 |
Viruspartikel (Virions) sind in der Regel Einheiten aus Nukleinsäure und Protein (Hüllprotein, Capsid), Viroide sind proteinlos. Von wenigen Ausnahmen abgesehen, sind Viren von keiner Membran umgeben: wo dies der Fall ist (Bild links), ist die Membran der Wirtszelle entnommen. Viren verfügen über keinen Energiestoffwechsel, sie sind also zu keinen synthetischen Leistungen befähigt und sind daher auch nicht in der Lage, sich selbst zu replizieren. Je nach Art der Wirtszellen wird zwischen Pflanzenviren, die sich nahezu ausschließlich in pflanzlichen Zellen vermehren, Bakterienviren (Bakteriophagen), die auf lebende Bakterienzellen angewiesen sind, und animalischen Viren (Tierviren) unterschieden.
|
Der Begriff Spezifität muß noch viel enger gefaßt werden, denn es gibt kein Pflanzenvirus schlechthin, sondern "Arten", wie z.B. das Tabakmosaikvirus (TMV), das in Nicotiana-Arten, manchen anderen Solanaceen und einigen wenigen Arten aus anderen Pflanzenfamilien vermehrbar ist. Meist ist die Bezeichnung für eine Virusart dem (englischen) Namen der Hauptwirtspflanze entlehnt. Obwohl die Bezeichnung "Art" vielleicht nicht ganz dem entspricht, was man sich in der biologischen Systematik unter dem Artbegriff vorstellt, ist es durchaus angebracht und üblich, ihn auf Viren auszudehnen, denn alle Viren (und Viroide) besitzen ein eigenständiges Genom, dessen Informationsgehalt artspezifisch ist und dessen Kontinuität über Generationen hinweg durch Replikation in den Wirtszellen gewährleistet ist. Die genetische Information der Viren ist entweder in einsträngiger RNS (bei den meisten Pflanzenviren), in doppelsträngiger RNS (Wundtumorvirus), einsträngiger DNS (Gemini-Viren) oder doppelsträngiger DNS (Cauliflower-Mosaik-Virus: CaMV) enthalten. Aufgrund der Form der Viruspartikel unterscheidet man zwischen den stäbchenförmigen und den icosaedrischen Viren, deren Capsid nahezu kugelig (sphärisch) erscheint.

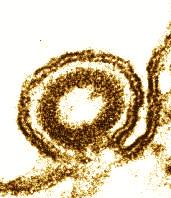
Tabakmosaikvirus: Elektronenmikroskopische Aufnahme - Präparationstechnik: negative staining. - Reliefstruktur durch Bildbearbeitung
Pflanzenviren verfügen über keinerlei spezifische Mechanismen, um in eine Wirtszelle einzudringen. Die Zellwände und die Kutikula sind für sie schwer zu überwindende Hindernisse. Sie sind daher auf Verletzungen oder auf eine Übertragung (Transmission) durch Invertebraten (Insekten, Nematoden u.a.) angewiesen. In einigen Fällen dienen die tierischen Überträger (Vektoren) auch als Zwischenwirte. Das wiederum heißt, daß sich einige Pflanzenviren auch in tierischen Geweben vermehren können.
Viruserkrankungen der Pflanzen sind relativ selten. Nur in Ausnahmefällen sind die Infektionen so stark, daß die Pflanze daran zugrunde geht. In Monokulturen ist die Ausbreitung naturgemäß begünstigt, und Ertragsminderungen in der Landwirtschaft (z.B. durch Kartoffel-X-, oder Kartoffel-Y-Virus) können merklich zu Buche schlagen.
Von vielen Viren sind zahlreiche Stämme (Wildstämme) isoliert worden, die sich beträchtlich voneinander unterscheiden. Zu den Unterschieden gehören der Wirtsbereich und das Ausmaß der Virulenz (= Schwere der Krankheitssymptome). Darüber hinaus sind Viroide (virusähnliche Einheiten) bekannt geworden. Bei ihnen handelt es sich um kleine zirkuläre RNS-Moleküle, die selbst keine Proteine codieren, die aber wegen ihrer Ähnlichkeit mit bestimmten Erkennungsregionen primärer Transkriptionsprodukte der Zellen in das Transkriptionsgeschehen eingreifen. Offensichtlich unterbinden sie das korrekte Ausschneiden der Introns. Vermehrt werden sie vermutlich durch Mitwirkung der zellulären DNS-abhängigen RNS-Polymerase II. Sie sind vornehmlich in wärmeren Gegenden verbreitet und richten als Erreger von Kartoffelkrankheiten oder der Cadang-Cadang-Krankheit der Palmen beträchtlichen wirtschaftlichen Schaden an.
Obwohl ein Virus, wie z.B. das TMV, die Wirtspflanze nicht gravierend beeinträchtigt, ist die Viruskonzentration in den Zellen beträchtlich. Oft enthalten sie voluminöse Viruskristalle. TMV war daher auch das erste Virus, das in großen Mengen und in reiner Form gewonnen werden konnte (W. M. STANLEY, 1935, seinerzeit Princeton University).
Pflanzen sind Virusinfektionen nicht schutzlos ausgesetzt. Nur einige wenige Virusarten sind in der Lage, in meristematisches Gewebe einzudringen oder eine Aufeinanderfolge von Pflanzengenerationen zu infizieren (vertikale Transmission). Ein wirkungsvoller Abwehrmechanismus ist die Hypersensitivität, die wir bereits im Zusammenhang mit Pilzinfektionen kennengelernt haben. Sie beruht auf dem Absterben von Zellen in unmittelbarer Nachbarschaft des primären Infektionsherds, wodurch ein Vordringen des Virus ins übrige Gewebe unterbunden wird. Die genetischen Grundlagen der Hypersensitivität sind bei Nicotiana tabacum analysiert worden. Es gibt ein Gen, dessen Genprodukt Hypersensitivität gegen alle TMV-Stämme gewährleistet; es gibt ein anderes, das sich nur gegenüber manchen TMV-Stämmen manifestiert. Das Symptom, das ein Virus am Infektionsherd hervorruft, wird Primärsymptom genannt; Symptome, die es durch Ausbreitung (Sekundärinfektion) in der ganzen Pflanze hinterläßt, heißen Sekundärsymptome.
Virusinfektionen sind meist an mosaikartig verteilten hellgrün-dunkelgrün-Mustern der Blätter erkennbar. Oft breitet sich eine Infektion - von den Blattadern ausgehend - über das ganze Blatt aus. Blätter, die während ihrer Entwicklung infiziert wurden, sind vielfach deformiert oder eingerollt.
Aufgehellte Blattbereiche, oft kreisförmig um den Infektionsherd ausgebildet, nennt man Chlorosen, abgestorbene Bereiche Nekrosen. Die Aufhellung beruht auf einem Chlorophyllabbau, als dessen Folge eine verringerte Photosyntheseleistung des Blattes zu verzeichnen ist. Starke Infektionen erkennt man an lokal vollständigem Chlorophyllverlust; die Bereiche sehen daher gelb aus, weil nur die Carotinoide erhalten bleiben. Manche der TMV-Stämme z.B. sind an solchen Symptomen erkennbar (Gelbstämme). Sie sind in der Natur sehr selten, weil eine derart starke Schädigung der Wirtspflanze auch ihre Replikations- und Ausbreitungschancen verringert.
Einige Viren vermehren sich in Pflanzen symptomlos, man spricht dann von latenter Infektion. Das Wundtumorvirus ruft die Bildung von Tumoren hervor. Das Krankheitsbild ist bei den meisten Viren gleichermaßen wirts- und virusspezifisch und stellt damit ein wichtiges diagnostisches Merkmal dar.
In einem 1934 durchgeführten Experiment zeigte G. SAMUEL, daß sich das TMV entlang der Leitbahnen der Pflanze ausbreitet, sich dabei sowohl des Auf- als auch des Abtransports bedient. Voll ausdifferenzierte Blätter und Wurzeln werden ebenso infiziert wie sich gerade entwickelnde Blätter.
Wegen der bereits erwähnten großen Mengen extrahierbaren Materials, sind Pflanzenviren (vor allem das TMV) seit Jahrzehnten bevorzugte Objekte der Grundlagenforschung. Das TMV war auch das erste elektronenmikroskopisch abgebildete biologische Objekt (KAUSCHE, G., PFANKUCH, E., RUSKA, A.; Berlin 1939).
Es gelang der Nachweis, daß isolierte TMV-RNS allein infektiös ist. Damit war zugleich der Beweis erbracht, daß auch RNS Träger genetischer Information sein kann (A. GIERER und G. SCHRAMM; Tübingen, 1956). Es war auch das erste Objekt, an dem demonstriert wurde, daß eine chemische Veränderung der RNS (Desaminierung von Adenin oder Cytosin nach Nitritbehandlung) mutagen ist (K. W. MUNDRY und A. GIERER, 1958) und daß sich derart erzeugte Mutationen im Hüllprotein manifestieren können (A. TSUGITA und H. FRAENKEL-CONRAT, Berkeley 1960; H. G. WITTMANN, Tübingen 1960).
Die Analyse einer großen Zahl chemisch induzierter Mutanten (durch die Arbeitsgruppen in Tübingen und Berkeley) brachte mit die ersten Ansätze zur Entschlüsselung des genetischen Codes. Die Ergebnisse WITTMANNs gaben Klarheit darüber, daß der genetische Code nicht überlappend und universell (d.h. gleichermaßen für Bakterien, Pflanzen und Tiere gültig) ist. Am Beispiel des Tabakmosaikvirus konnte ferner demonstriert werden, daß die gentragende RNS nicht zugleich als mRNS fungiert; vielmehr wird diese als eine getrennte Fraktion in der Zelle synthetisiert (T. R. HUNTER et al., Cambridge 1976).
Die Arbeitsgruppe um A. KLUG (Medical Research Council, Laboratory of Molecular Biology, Cambridge) befaßt sich seit über zwei Jahrzehnten mit der Frage nach dem Assembly (der Zusammenlagerung) der Proteinuntereinheiten zu kompletten Viruspartikeln und der Integration der RNS in die sich bildende Helix aus Proteinuntereinheiten.
Die Fortentwicklung der Röntgenstrukturanalyse und die schnellere Auswertung der Meßdaten durch Einsatz einer neuen Computergeneration erlaubte die Aufklärung der Proteintertiärstruktur. Durch konsequente Weiterentwicklung der hier erarbeiteten Versuchsansätze konnten S. HARRISON und Mitarbeiter (Harvard University) zu Beginn der achtziger Jahre eine Anzahl sphärischer Viren analysieren und deren Tertiär- und Quartärstrukturen aufklären. Die Auflösungsgrenze (2,8-2,9Å) ist ausreichend, um einzelne Aminosäurereste lokalisieren zu können.
Eine wesentliche Erkenntnis ist der Nachweis der sogenannten semi-äquivalenten Aggregation. Das Hüllprotein dieser Viren kann sich zu unterschiedlichen Formen, die sich durch die Zahl der Symmetrieachsen und Nachbarschaftbeziehungen voneinander unterscheiden, zusammenlagern. Die Zusammenlagerung dieser Aggregate erklärt den Aufbau so komplexer Strukturen wie die einer Hohlkugel. Die Untersuchungen haben zugleich Modellcharakter, denn komplexe Strukturen dieser Art sind in Zellen nicht selten. Ein Beispiel: Die korkenzieherartig gebaute Geißel der Prokaryoten ist ein Aggregat aus Molekülen eines einheitlichen Typs (Flagellin).
Warum haben sich die Molekularbiologen trotz der eben skizzierten - und doch wohl eindrucksvollen - Vorteile der Pflanzenviren, jahrzehntelang mit Bakteriophagen (Phagen) befaßt? Auch hierauf gibt es plausible Antworten:
Der Vermehrungsmechanismus der Phagen ist leichter faßbar. Ihre Wirtszellen, die Bakterien (am besten untersucht ist Escherichia coli) sind leichter kultivierbar, die Generationszeiten sind kurz. Ein vollständiger Vermehrungszyklus der Phagen ist in ca. 15 Minuten abgeschlossen. Das System Phage/Bakterium ist weniger komplex als das Virus/Pflanzen-System. Die einzelnen Parameter sind daher leichter meßbar. Der Infektionsvorgang ist ein Eintrefferprozeß, im Falle der Pflanzenviren ist es oft ein Vieltrefferprozeß (Multitrefferprozeß).
Bakterien- und Bakteriophagengenetiker waren in erster Linie an der (doppelsträngigen) DNS der Phagen interessiert (es gibt auch RNS-haltige und Einstrang-DNS-haltige Phagen). In DNS-Doppelstrang gespeicherte Information kann rekombiniert werden, die Anordnung der Gene ist bestimmbar. Es gibt keine Rekombinationsereignisse bei RNS, und es ist daher sehr schwierig, etwas über die Genomorganisation der Pflanzenviren auszusagen. Mit den modernen Methoden der Gentechnik eröffneten sich inzwischen aber auch hier Wege, um ans Ziel zu gelangen. Als Nachteil der Phagen sei die meist komplexe Struktur ihrer Capside genannt. Die Hülle ist oft aus mehr als 20 voneinander verschiedenen Proteinen zusammengefügt.
Das Studium der Bakterien und ihren Bakteriophagen brachte zahlreiche Erkenntnisse über den Infektionsverlauf und die Biosynthese dieser Viren. Aus Untersuchungen dieser Art lernte man, zu welchen Leistungen eine Zelle befähigt ist. So wurde u.a. Ende der fünfziger Jahre die mRNS in einem phageninfizierten Bakterium entdeckt. Anschließend erkannte man, daß sie ein essentielles Zwischenprodukt einer jeden Proteinbiosynthese ist.
Durch das Studium phytopathogener Viren erhofft man sich - auf den Untersuchungen an Bakterien aufbauend - Angaben über die Möglichkeiten und die Expression des pflanzlichen Genoms sowie über Abwehrmechanismen einer Pflanze gegenüber eingebrachter Fremd-RNS oder -DNS.
Auf den Ergebnissen der Molekularbiologie, speziell der Virusforschung der letzten Jahrzehnte aufbauend, bemüht man sich mit Hilfe nunmehr verfügbarer gentechnischer Methoden einen Schutz vor Virusinfektionen zu erzielen. Ein Beispiel hierfür ist der Ansatz zur Induktion eines Abwehrmechanismus gegenüber dem Blattroll-Virus. Ein Bericht von W. SCHUCHERT, E. PEERENBOOM und MPI für Züchtungsforschung hierzu findet man unter
Die virusresistente Kartoffel - Gentechnisch vermittelter Breitbandschutz vor den schädlichsten Viren der Kartoffel
|
|